Autor: Dr. Elias Puchner
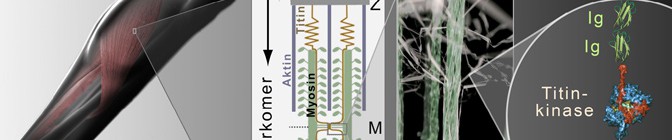
In dieser Arbeit wurde die Funktion und der molekulare Aktivierungsmechanismus des autoinhibierten Muskelenzyms Titinkinase in der natürlichen biomolekularen Umgebung mit Einzelmolekül-Kraftspektroskopie entschlüsselt.
Die entwickelte Basistechnologie erlaubt die Nanoassemblierung verschiedenster funktioneller Einheiten und könnte die Konstruktion von synergetischen Enzymkaskaden, oder das Imitieren biologischer Signalwege ermöglichen.
Es konnte gezeigt werden, dass die Titinkinase über fünf Energiebarrieren und mit Kräften unter 50 pN vor den strukturell wirkenden Titindomänen (Ig/Fn) entfaltet. In Anwesenheit des Cosubstrates ATP wurde eine zusätzliche Energiebarriere beobachtet. Die präzise Lokalisierung der Energiebarrieren bis auf wenige Aminosäuren genau erlaubte die Bestimmung aller strukturellen Veränderungen während der Entfaltung.
Neu entwickelte mechanische „pump-and-probe“-Protokolle erlaubten die detaillierte Vermessung der ATP-Bindungskinetik, die Lokalisierung der mechanischen Aktivierung nach der zweiten Energiebarriere und wiesen die essenzielle Rolle der Aminosäure Lysin-36 für die ATP-Bindung nach.Die Titinkinase startet katalytisch aktiv die Signalkaskade zur Regulation von Muskel-Genexpression und Proteinumsatz. Sie wirkt als natürlicher Kraftsensor, um den Muskelaufbau zu steuern.
Das Konzept der mechanisch induzierten Konformationsänderung wurde mittels TIRF-Mirkoskopie und Substratzugabe auch auf die Lipase CalB übertragen. Zur kontrollierten Oberflächenassemblierung wurde mittels der „Single-Molecule Cut-and-Paste“-Technik das Prinzip der Selbstorganisation mit der Präzision des Atomic Force Microscope (AFM) kombiniert.
Gesamte Publikation lesen
Disertation: Einzelmolekuel-Mechanoenzymatik >>
Zur Autoren-Homepage
Dr. Elias Puchner >>

One thought on “Einzelmolekül – Mechanoenzymatik”
Kommentare sind geschlossen.